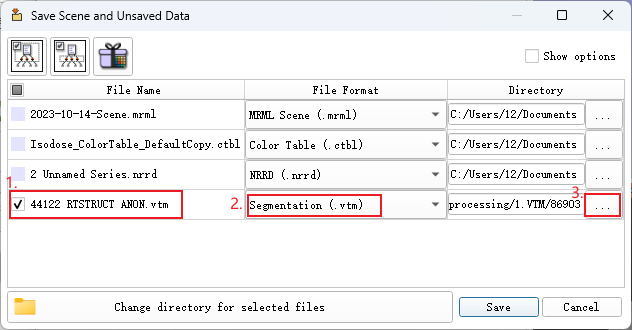
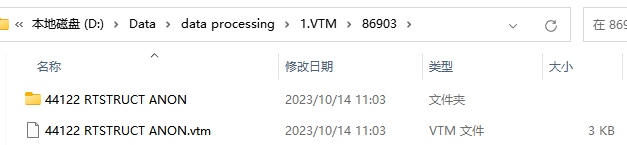
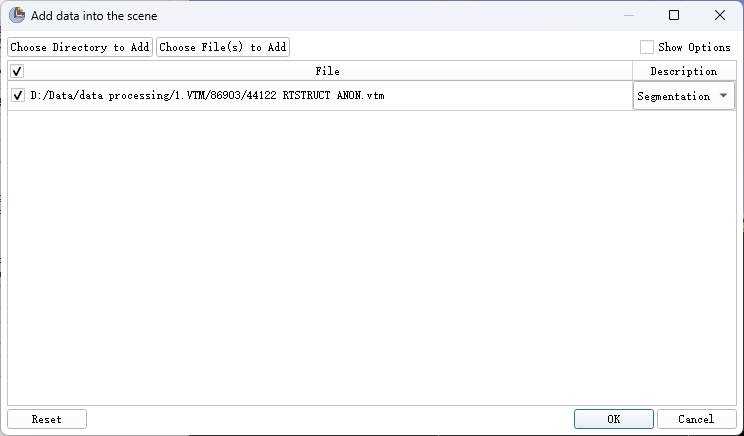
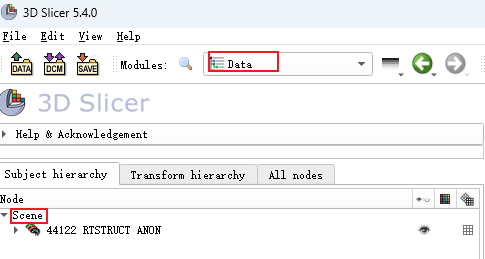
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
| from PIL import Image
import numpy as np
import SimpleITK as sitk
import os
base_folder = r"D:\Data\data processing"
bmp_folser = os.path.join(base_folder, "3.bmp_new")
subfolders = [f for f in os.listdir(bmp_folser) if os.path.isdir(os.path.join(bmp_folser, f))]
for subfolder in subfolders:
print(f"Processing folder: {subfolder}")
input_folder_bmp = os.path.join(base_folder, f"3.bmp_new\{subfolder}")
output_folder_bmp = os.path.join(base_folder, f"3.bmp2\{subfolder}")
output_folder_nii = os.path.join(base_folder, f"4.NII\{subfolder}")
output_file_label = os.path.join(base_folder, f"5.Label_new\{subfolder}_label.nii")
try:
os.makedirs(output_folder_bmp, exist_ok=True)
os.makedirs(output_folder_nii, exist_ok=True)
bmp_files = [f for f in os.listdir(input_folder_bmp) if f.endswith('.bmp')]
for bmp_file in bmp_files:
bmp_path = os.path.join(input_folder_bmp, bmp_file)
try:
image = Image.open(bmp_path)
new_size = (512, 512)
image = image.resize(new_size, Image.LANCZOS)
image = image.convert('1')
output_path_bmp = os.path.join(output_folder_bmp, os.path.splitext(bmp_file)[0] + '_1bit.bmp')
image.save(output_path_bmp)
bmp_image = np.array(image)
bmp_image = bmp_image.astype(np.uint8)
sitk_image = sitk.GetImageFromArray(bmp_image)
output_file_nii = os.path.join(output_folder_nii, os.path.splitext(bmp_file)[0] + '.nii')
sitk.WriteImage(sitk_image, output_file_nii)
except Exception as e:
print(f"Error processing file {bmp_file}: {e}")
nii_files = [os.path.join(output_folder_nii, f) for f in os.listdir(output_folder_nii) if f.endswith('.nii')]
sitk_images = []
for nii_file in nii_files:
try:
sitk_image = sitk.ReadImage(nii_file)
sitk_images.append(sitk_image)
except Exception as e:
print(f"Error reading NII file {nii_file}: {e}")
output_image = sitk.JoinSeries(sitk_images)
desired_spacing = [1.03711, 1.03711, 5.0]
desired_origin = [-265.5, -207.5, -833.0]
output_image.SetSpacing(desired_spacing)
output_image.SetOrigin(desired_origin)
output_label_path = os.path.join(base_folder, f"5.Label\{subfolder}_label.nii")
sitk.WriteImage(output_image, output_label_path)
print(f"Processing completed for folder: {subfolder}")
except Exception as ex:
print(f"Error processing folder {subfolder}: {ex}")
print("All folders processed.")
|